科研服务
立足于生命科学,为基础研究领域科学工作者提供生物学高端技术服务
科研服务
相关产品

- 产品描述
-
服务介绍
LC-MS/MS代谢组学由于其灵敏度高、通用性强、无需化学衍生等优点,近年来得到了广泛的应用。基于高分辨LC-MS/MS非靶向代谢组学平台,对生物样本中提取到的代谢小分子化合物进行大规模、无偏向性检测,对代谢物的质谱峰强度值进行定量分析,将代谢物的碎片图谱与本地标准品二级谱图数据库进行匹配得到物质鉴定结果。
应用领域
农业研究:用于植物抗逆、育种及有效成分研究。
微生物组-宿主互作研究:研究宿主与微生物共同参与代谢的产物。
药物作用机制研究:系统性揭示药物处理后细胞内/生物体内发生的代谢通路变化,从而推断其作用机制。
技术优势
鉴定数量提升:平均定性数量1500种左右,最高检测能力可到4000+,比常规代谢组提高2倍以上。
优质数据库:具有出色的代谢物覆盖种类和数量,约3万+的高质量标准品二级谱库。
高标准:保留高Similarity Score的谱图匹配结果,所有定性的代谢物均可提供二级匹配结果。
高精准:通过上百种混合标准品样本对定性准确率进行测评,定性准确高。
服务流程
主要步骤包括:样品制备、QC 制备、样品 LC-MS/MS 质谱分析、数据分析和实验报告等。
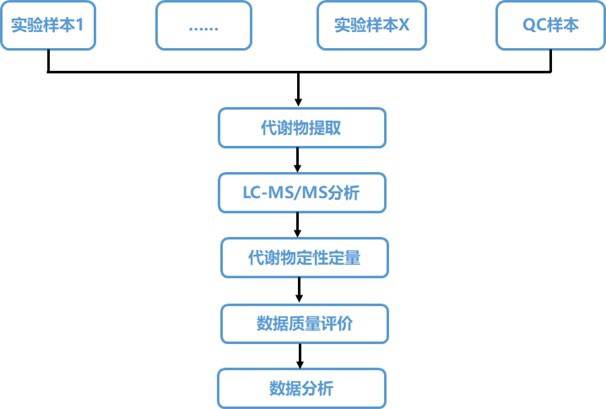
注:QC样本是由待测样本的提取液等量混合而成,在待测样本LC-MS/MS进样前、进样中和进样后上机检测。
样品接收标准
样本
要求
备注
植物组织
≥400 mg,≥6个生物学重复
液氮速冻,-80℃保存,干冰运输,1天(5kg)2-3天(10-20kg)3-5天(20-30kg)
动物组织 ≥400 mg,≥10个生物学重复 细胞类 ≥5x107 个,6个生物学重复 菌体 ≥400 mg,≥6个生物学重复 服务流程、周期及交付
服务流程
工作日
交付材料
样品制备
28个
- 原始数据、结果图片
- 标准实验报告(含实验步骤、流程)
3.生信分析:代谢物鉴定及分类、差异代谢物筛选、差异代谢物表达分析、差异代谢物功能分析
QC制备 LC-MS/MS质谱分析 生信分析 实验报告 数据库预览
类别
数量
代表性物质概览
碳水化合物及衍生物
1000+
葡萄糖、6-磷酸葡萄糖、果糖、6-磷酸果糖、核糖、5-磷酸核糖...
氨基酸及衍生物
2900+
谷氨酸、色氨酸、甘氨酸、赖氨酸、精氨酸、异亮氨酸、天冬氨酸...
有机酸及衍生物
650+
乳酸、尿素、柠檬酸、琥珀酸、苹果酸、马来酸、延胡索酸...
核苷酸、辅酶及衍生物
200+
ATP、UTP、AMP、GMP、CMP、TMP、NAD+、辅酶A、辅酶Q1、辅酶Q10...
维生素及衍生物
60+
维生素C、维生素D、维生素K、维生素B、维生素E、叶酸...
胆汁酸、胆汁醇及衍生物
320+
胆酸、猪胆酸、甘胆酸、甘氨脱氧胆酸、石胆酸、葡糖石胆酸...
固醇及类固醇
1700+
胆固醇、孕烷类固醇、雄烷类固醇、雌烷类固醇...
类花生酸
380+
前列腺素类、白三烯类、5-羟基二十碳五烯酸(HEPE)、二十烷四烯酸(HETE)...
脂质
4800+
甘油三脂、甘油二脂、亚油酸、神经酰胺、甘油磷脂、甘油磷酸脂...
短肽
1900+
二肽、三肽、四肽...
常见生物碱
110+
咖啡因、吗啡、茶碱、麻黄碱、烟碱、尼古丁...
苯及其衍生物
3100+
苯甲酸、本甲酰胺、马尿酸、4-羟基苯甲酸、3,4-二甲氧基苯乙酸...
杂环化合物
3900+
核酸、维生素、激素、色素和生物碱等代谢物的前体及衍生物
醇、胺
2100+
多巴胺、组胺、精胺、组氨醇、肌醇...
酮、醛、酯、酚
3200+
丙酸乙脂、乙酸乙脂、肉桂醛...
其他
3700+
常见环境污染物及药物
数据分析
服务标准
分析类型
分析内容
拟解决问题
标准数据分析
质控
TIC等多项QC分析
获得高质量数据
轮廓分析
代谢物化学分类归属统计
获得代谢物的分类信息
差异分析
单变量统计分析(火山图等)
反映每个变量的组间差异
多维统计分析(PCA、PLS-DA、OPLS-DA等)
评估组间差异,找到导致组间差异的影响变量
差异倍数分析(蝴蝶图)
直观展示差异代谢差异倍数变化
韦恩分析
展示不同对比组中共有和特有的差异代谢物
丰度分析(柱状图、箱线图、小提琴图)
展示每个物质最小值、最大值、平均水平等信息
层次聚类分析
获得代谢物的表达模式信息
相关性分析(相关性热图、和弦图、网络图)
衡量两个变量的相关程度
功能分析
KEGG注释分析
进一步了解相关代谢通路和生物学功能
KEGG通路富集分析(气泡图、得分图、网络图等)
筛选重要通路
MetPA富集分析
筛选重要通路
MSEA富集分析
筛选重要通路
诊断分析
ROC分析
表明代谢物区分两组间的能力
高级数据分析
生信分析
WGCNA
获得与表型强关联的核心代谢物
与目标参数相关的代谢物筛选
OPLS回归分析
临床指标关联分析
建立重要代谢物和临床指标的关联
分子分型分析
基于代谢物表达趋势分对样本分类
机器学习分析
筛选诊断性能良好的生物标志物
两组学分析
转录组/蛋白组/代谢组联合分析
三组学分析
转录组+蛋白组+代谢组联合分析
案例报告
1、鉴定结果统计
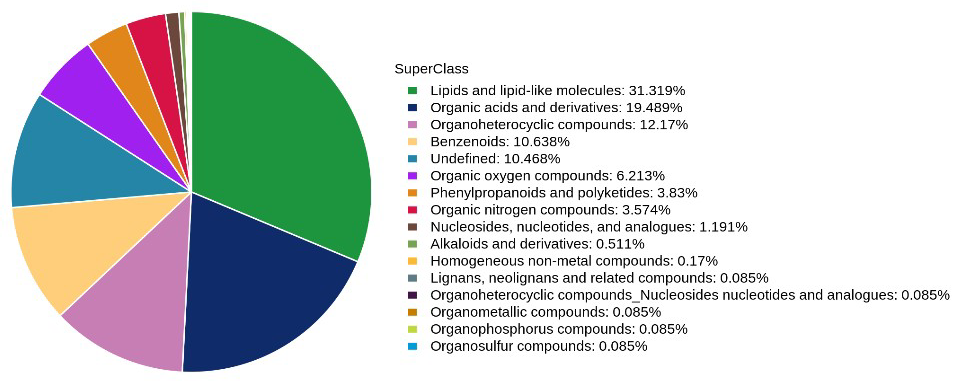
图:鉴定的代谢物在各化学分类的数量占比
2、多维统计分析
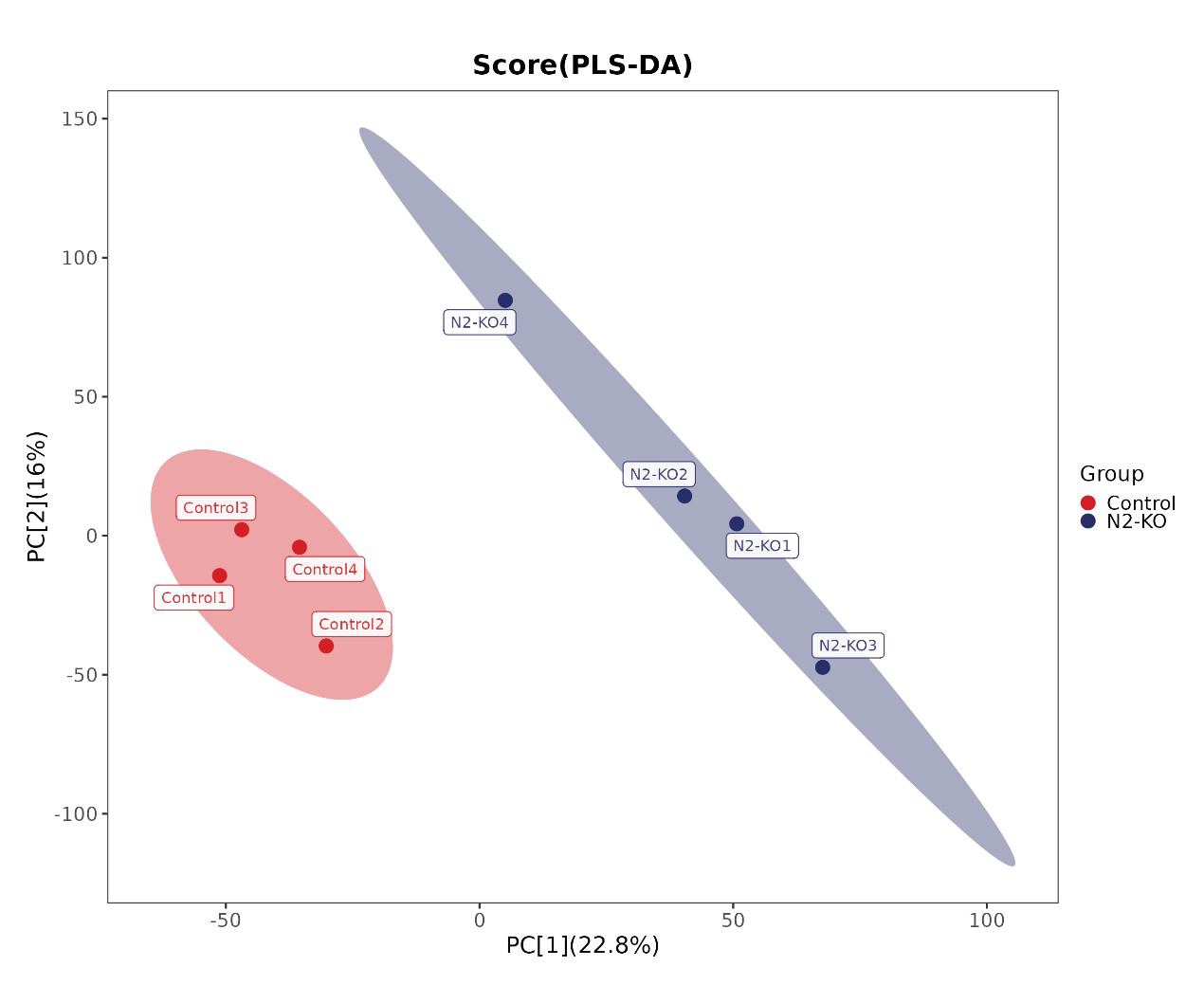
图:PLS-DA得分图
3、差异分析
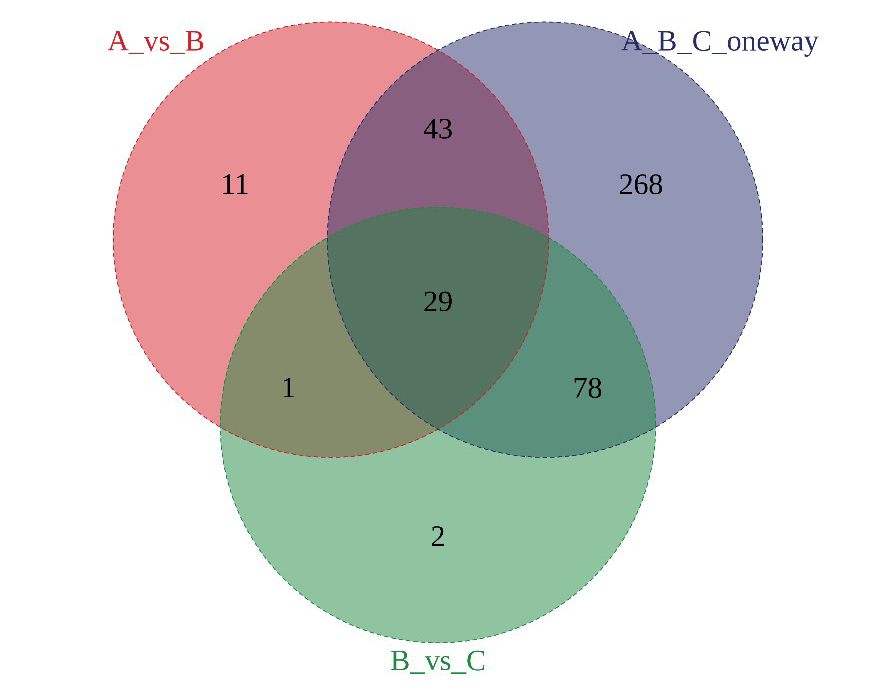
图:差异代谢物韦恩图
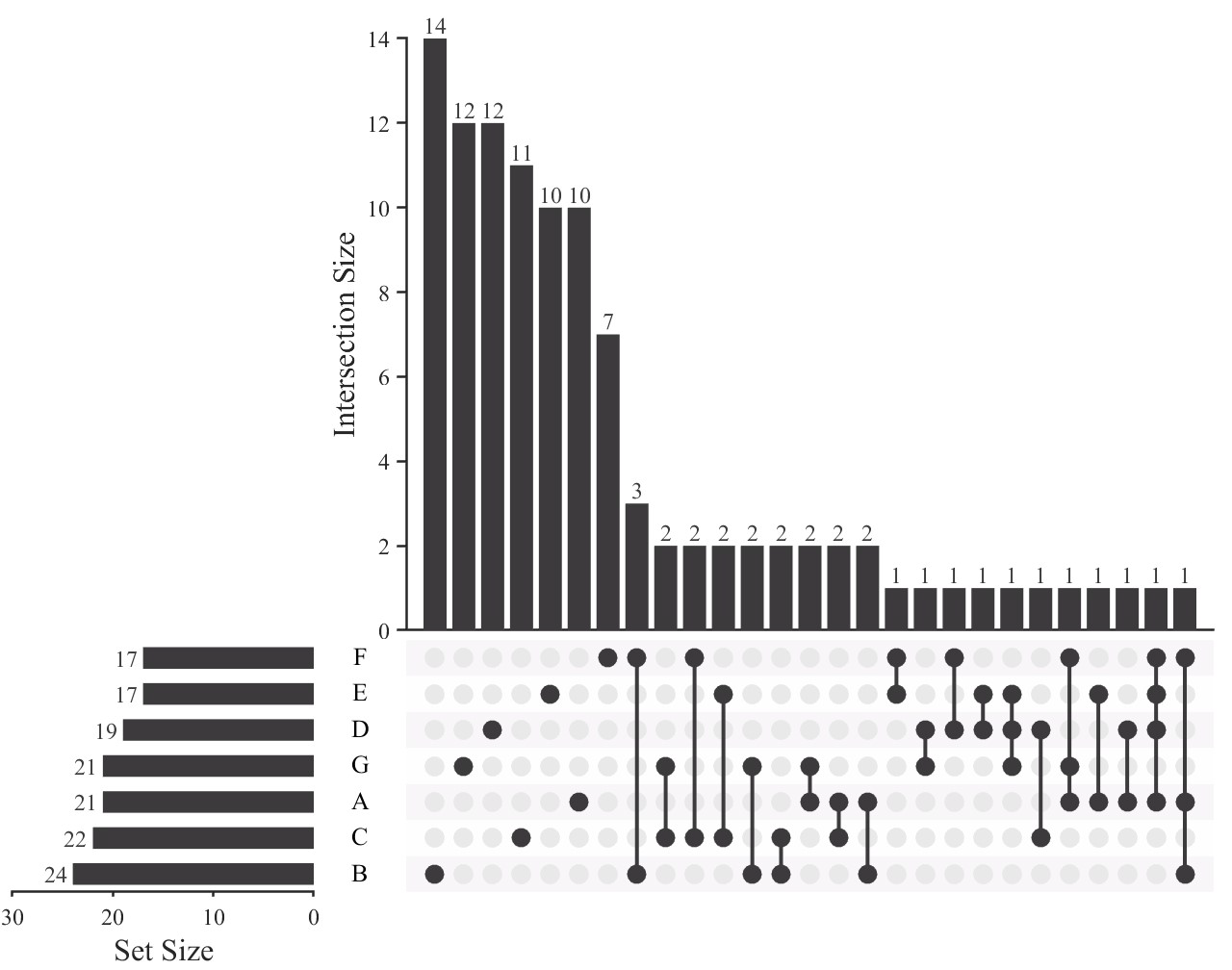
图:差异代谢物Upset图
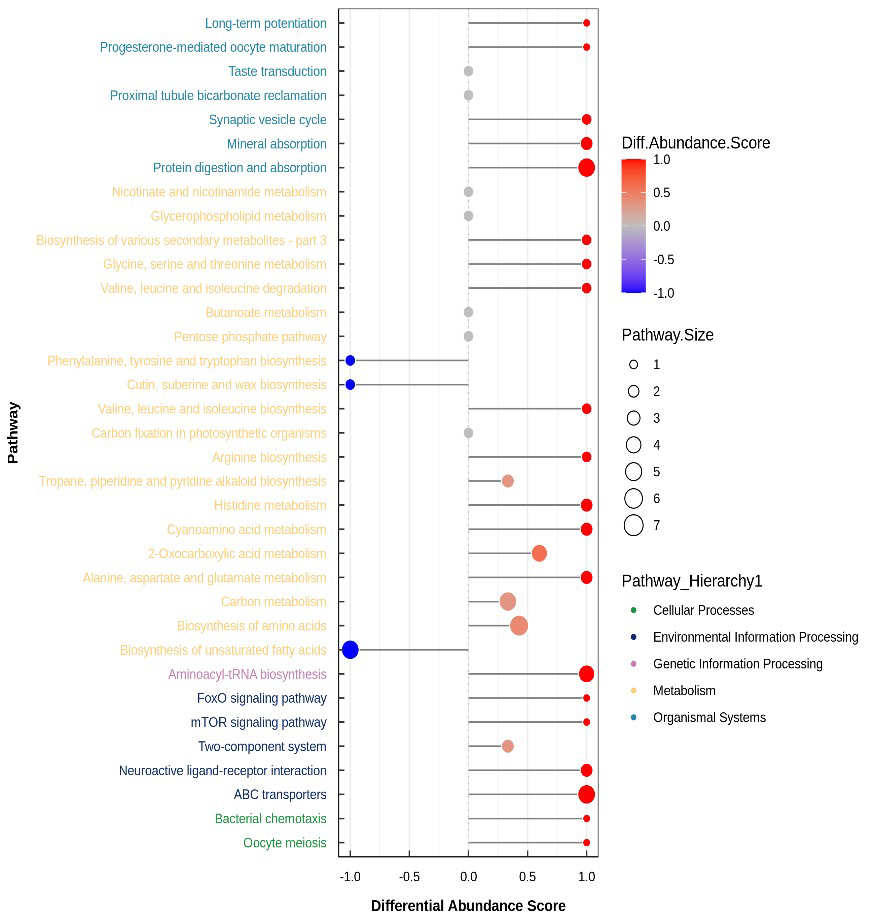
图:所有富集代谢通路的差异丰度得分图(按照Pathway_Hierarchy进行分类归属)
4、MSEA富集分析
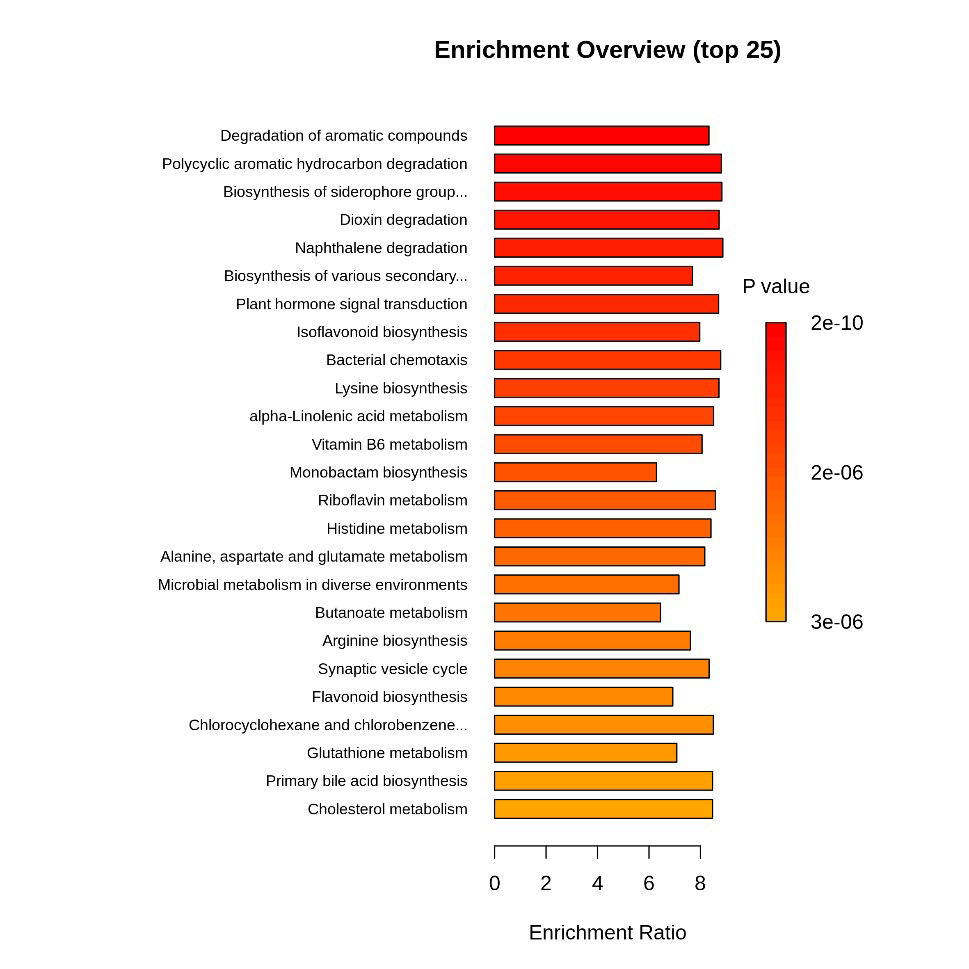
图:MSEA富集分析图
5、ROC分析
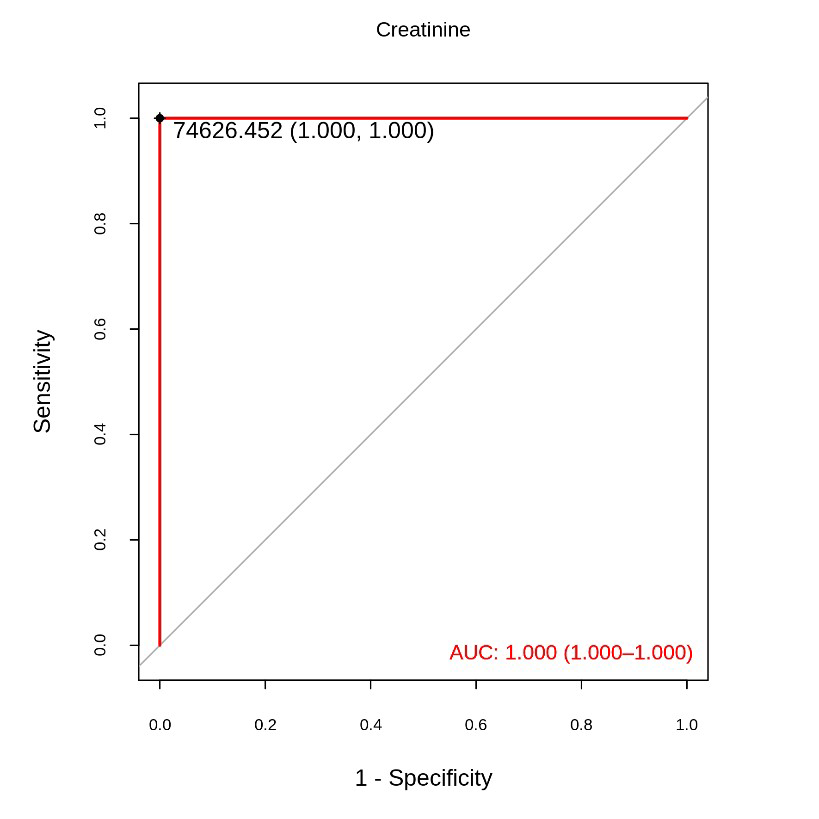
图:ROC分析
6、高级分析(仅做展示,需要请另询)
图:WGCNA聚类树状图
参考文献
[1] Wu RQ, Zhao XF, Wang ZY, et al. Novel molecular events in oral carcinogenesis via
integrative approaches. J Dent Res. 2011 May;90(5):561-72.
[2] Nicholson JK, Lindon JC. Systems biology: Metabonomics. Nature. 2008 Oct
23;455(7216):1054-6.
[3] Zhang XJ, Liu X, Hu M, et al. Pharmacological inhibition of arachidonate 12lipoxygenase ameliorates myocardial ischemia-reperfusion injury in multiple species. Cell Metab. 2021 Oct 5;33(10):2059-2075.e10.
[4] Cong J, Liu P, Han Z, et al. Bile acids modified by the intestinal microbiota promote
colorectal cancer growth by suppressing CD8+ T cell effector functions. Immunity. 2024
Apr 9;57(4):876-889.e11.
[5] Blaženović I, Kind T, Ji J, et al. Software Tools and Approaches for Compound
Identification of LC-MS/MS Data in Metabolomics. Metabolites. 2018May 10;8(2):31.
[6] Qi Z, Zhao R, Xu J, et al. Accumulation Pattern of Flavonoids during Fruit Development of Lonicera maackii Determined by Metabolomics. Molecules. 2021 Nov
16;26(22):6913.(QC TIC).
[7] Barker-Tejeda TC, Zubeldia-Varela E, Macías-Camero A, et al. Comparative characterization of the infant gut microbiome and their maternal lineage by a multi-omics approach. Nat Commun. 2024 Apr 8;15(1):3004.(QC-PCA).
[8] Siskos AP, Jain P, Römisch-Margl W, et al. Interlaboratory Reproducibility of a Targeted Metabolomics Platform for Analysis of Human Serum and Plasma. Anal Chem. 2017 Jan 3;89(1):656-665.
[9] Shen, X., Gong, X., Cai, Y. et al. Normalization and integration of large-scale metabolomics data using support vector regression. Metabolomics. 12, 89 (2016).
[10] Blaženović I, Kind T, Ji J, Fiehn O. Software Tools and Approaches for Compound Identification of LC-MS/MS Data in Metabolomics. Metabolites. 2018 May 10;8(2):31.
[11] Contrepois K, Wu S, Moneghetti KJ, et al. Molecular Choreography of Acute Exercise. Cell. 2020 May 28;181(5):1112-1130.e16.
[12] Liang L, Rasmussen MH, Piening B, et al. Metabolic Dynamics and Prediction of Gestational Age and Time to Delivery in Pregnant Women. Cell. 2020 Jun 25;181(7):1680-1692.e15.
[13] Luo D, Deng T, Yuan W, et al. Plasma metabolomic study in Chinese patients with wet age-related macular degeneration. BMC Ophthalmol. 2017 Sep 6;17(1):165.
[14] Gu Z, Li L, Tang S, et al. Metabolomics Reveals that Crossbred Dairy Buffaloes Are More Thermotolerant than Holstein Cows under Chronic Heat Stress. J Agric Food Chem. 2018 Dec 12;66(49):12889-12897.
[15] Gao Yawei, Liu Xiaoyu, Tang Bin et al. Protein Expression Landscape of Mouse Embryos during Pre-implantation Development. Cell Rep, 2017, 21: 3957-3969.
[16] Takefuji Y. Revealing Bias in Feature Importance through PLS-DA: A Critical Examination of Machine Learning Applications in Chronic Liver Disease. J Hepatol. 2024 Dec 17:S0168-8278(24)02776-4.
[17] Sun X, He L, Liu H, et al. The diapause-like colorectal cancer cells induced by SMC4 attenuation are characterized by low proliferation and chemotherapy insensitivity. Cell Metab. 2023 Sep 5;35(9):1563-1579.e8.
[18] Atrahimovich D, Khatib S, Sela S, et al. Punicalagin Induces Serum Low-Density Lipoprotein Influx to Macrophages. Oxid Med Cell Longev. 2016;2016:7124251.
[19] Chen X, Wei J, Li Z, et al. Dysregulation of Gut Microbiota-Derived Neuromodulatory Amino Acid Metabolism in Human Immunodeficiency Virus-Associated Neurocognitive Disorder: An Integrative Metagenomic and Metabolomic Analysis. Ann Neurol. 2024 Aug;96(2):306-320.
[20] Prins FM, Collij V, Groot HE, et al. The gut microbiome across the cardiovascular risk spectrum. Eur J Prev Cardiol. 2024 Jun 3;31(8):935-944.
[21] Xia J, Wishart DS. MetPA: a web-based metabolomics tool for pathway analysis and visualization. Bioinformatics. 2010 Sep 15;26(18):2342-4.
[22] Xia J, Wishart DS. MSEA: a web-based tool to identify biologically meaningful patterns in quantitative metabolomic data. Nucleic Acids Res. 2010 Jul;38(Web Server issue):W71-7.
[23] Jia P, Wang Q, Chen Q, et al. MSEA: detection and quantification of mutation hotspots through mutation set enrichment analysis. Genome Biol. 2014;15(10):489.
[24] Mandrekar JN. Receiver operating characteristic curve in diagnostic test assessment. J Thorac Oncol. 2010 Sep;5(9):1315-6.
[25] Yu H, Du X, Zhao Q, et al. Weighted gene Co-expression network analysis (WGCNA) reveals a set of hub genes related to chlorophyll metabolism process in chlorella (Chlorella vulgaris) response androstenedione. Environ Pollut. 2022 Aug 1;306:119360.
[26] Baggerly KA, Morris JS, Edmonson SR, Coombes KR. Signal in noise: evaluating reported reproducibility of serum proteomic tests for ovarian cancer. J Natl Cancer Inst. 2005 Feb 16;97(4):307-9.
- 原始数据、结果图片
声明:本公司出售产品只能用于科研目的,不得用于诊断或者治疗!


