科研服务
立足于生命科学,为基础研究领域科学工作者提供生物学高端技术服务
科研服务
相关产品

- 产品描述
-
服务介绍
4D蛋白质组学是基于新一代timsTOF Pro2质谱仪,通过捕集离子淌度(TIMS)与同步累积连续碎裂(PASEF)扫描模式进行差异定量蛋白质组学研究。以更少上样量、更快扫描速度,实现蛋白质组、修饰组学在覆盖深度、灵敏度、通量方面的全面性提升。
应用领域
农业研究:用于作物表型性状与蛋白表达的关联研究。
药物靶点发现:筛选疾病关键蛋白、作用机制相关蛋白。
机制研究:分析代谢通路、信号通路的动态调控变化。
蛋白表达定量分析:高通量检测样本中蛋白质表达水平变化。
技术优势
专业设备:基于新一代timsTOF Pro2质谱仪,检测速度提升近一倍,通过捕集离子淌(TIMS)与同步累积连续碎裂(PASEF)扫描模式进行差异定量蛋白质组学研究;
高精准:利用第四维“离子淌度”,有效区分同分异构肽段,显著提升鉴定准确性;
高深度:蛋白鉴定水平提升近20%,具有更高的检测深度和覆盖度;
服务流程
样品接收标准
样本
要求
备注
植物组织
≥200 mg
液氮速冻,-80℃保存,干冰运输,1天(5kg)2-3天(10-20kg)3-5天(20-30kg)
动物组织
≥100 mg
细胞类
≥5 x 107 个
菌体
≥100 mg
服务流程、周期及交付
服务流程
工作日
交付材料
蛋白提取及处理
18个
1、原始数据、结果图片
2、标准实验报告(含实验步骤、流程)
3、生信分析:鉴定数量分析、表达差异分析、功能分析
DIA LC-MS/MS
数据库检索
定性定量分析
生信分析
实验报告
数据分析
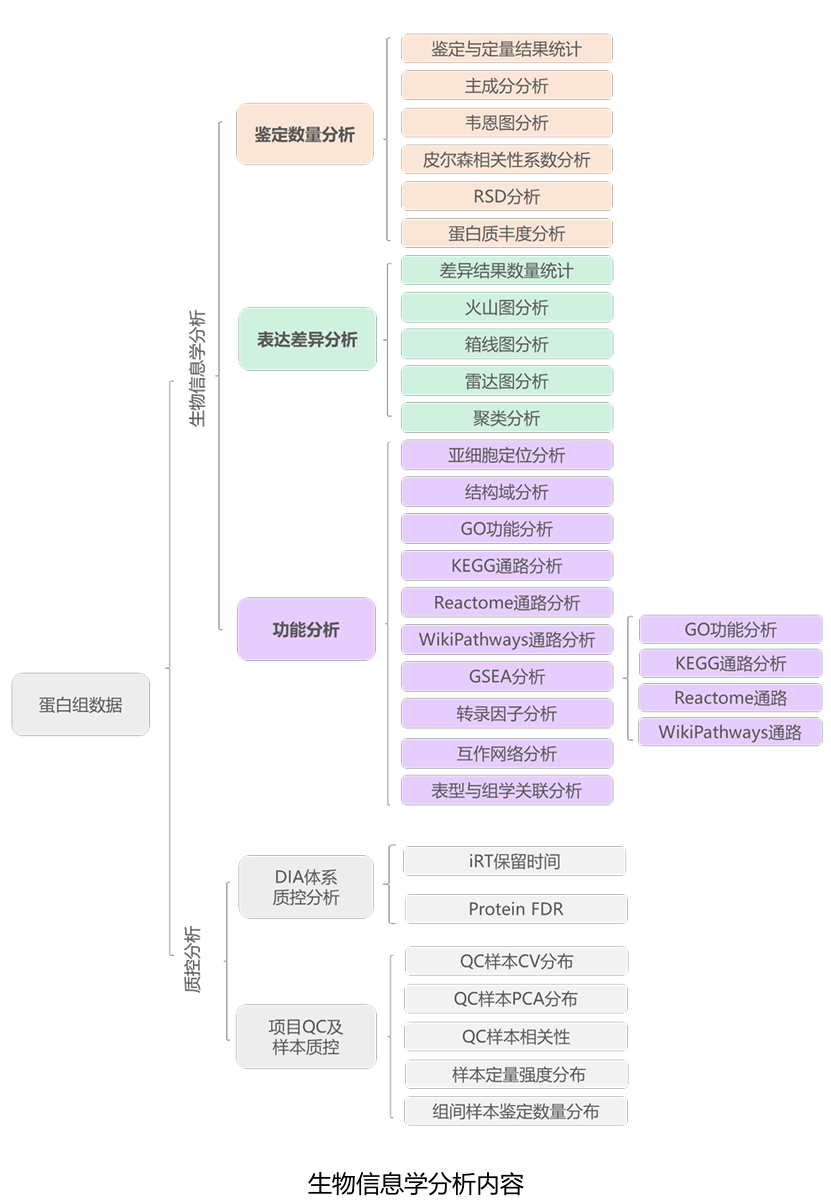
标准数据分析内容
鉴定分析 鉴定与定量结果统计;单个样本蛋白质鉴定结果统计;韦恩图;主成分分析;PCC分析;RSD分析;蛋白质丰度分析 差异表达分析 差异结果统计;火山图;箱线图;雷达图;聚类分析图 功能分析 亚细胞定位;结构域分析;GO功能分析;KEGG/Reactome/ WikiPathways通路分析;GSEA分析;转录因子分析;激酶分析;互作网络分析;WGCNA分析 质量控制 QC样本评价;组间样本鉴定评价;样本定量波动评价;DIA体系评价 高级数据分析 临床药物研究 药物靶点注释 标志物的筛选 集成机器学习 分子分型 无监督聚类分型分析 案例报告
1、鉴定数量分析
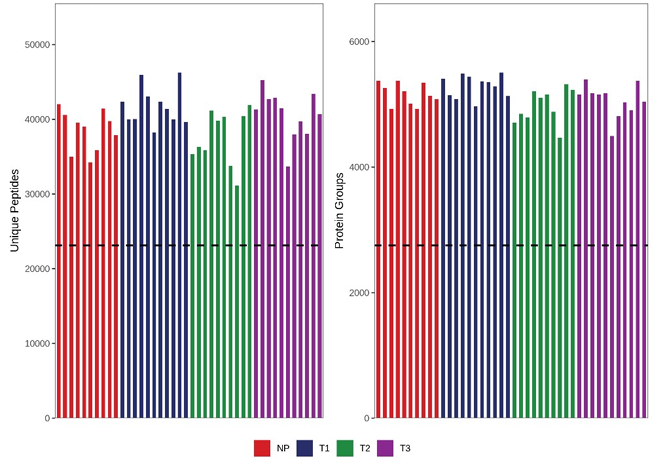
图:DIA 鉴定结果统计柱状图
2、表达差异分析
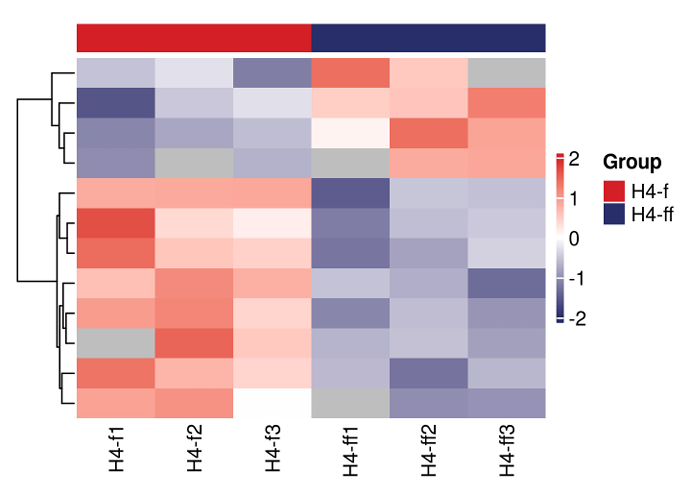
图:groupvs 组差异表达蛋白质聚类分析图
3、功能分析
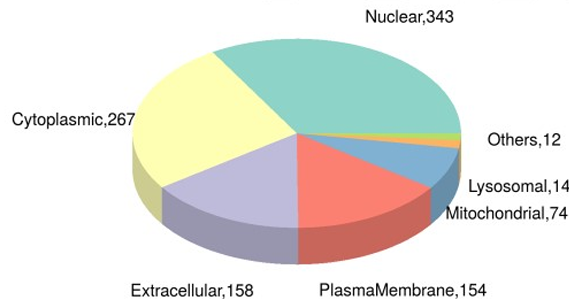
图:groupvs 组差异表达蛋白质亚细胞定位分布饼图
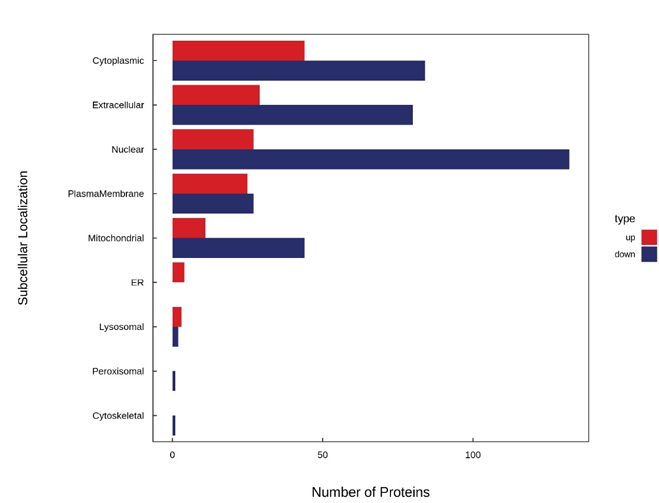
图:亚细胞定位结果上下调比较柱状图
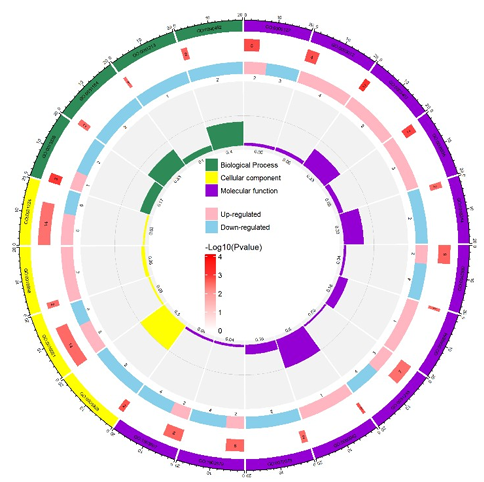
图:groupvs 组所有差异蛋白质的 GO 富集 Circos
3、质量控制(QC)
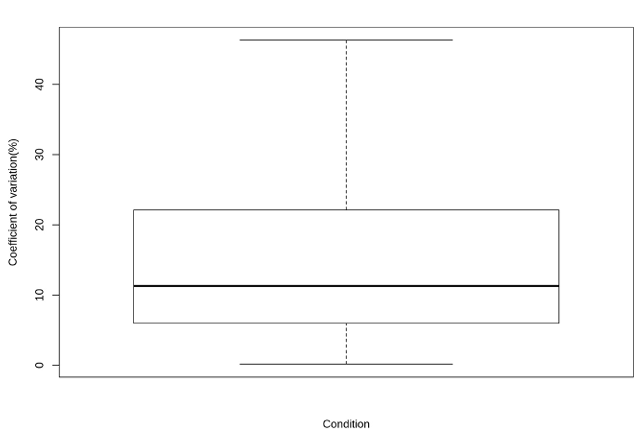
图:QC 的 CV 值分布箱线图
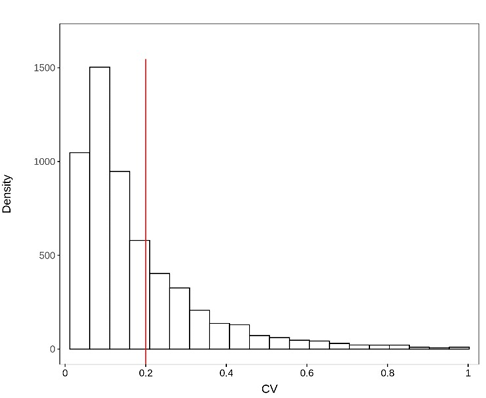
图:QC 的 CV 值分布图
图:QC 样本的3D-PCA 图
参考文献
[1] Yu CS1, Lin CJ, Hwang JK. Predicting subcellular localization of proteins for Gram-negative bacteria by support vector machines based on n-peptide compositions. Protein Sci. 2004 May;13(5):1402-6.
[2] Finn RD, Coggill P, et al., The Pfam protein families database: towards a more sustainable future. Nucleic Acids Res. 2016 Jan 4;44(D1):D279-85.
[3] Ashburner, M., et al., Gene ontology: tool for the unification of biology. The Gene Ontology Consortium. Nat Genet, 2000. 25(1): p.25-9.
[4] Gotz, S., et al., High-throughput functional annotation and data mining with the Blast2GO suite. Nucleic Acids Res, 2008. 36(10): p. 3420-35.
[5] Kanehisa, M., et al., KEGG for integration and interpretation of large-scale molecular data sets. Nucleic Acids Res, 2012. 40(Database issue): p. D109-14.
[6] Subramanian,A.,et al.,. Gene set enrichment analysis: A knowledge-based approach for interpreting genome-wide expression profiles. Proc Natl Acad Sci U S A.,2005, 102(43):15545-50.
[7] Parker SJ, Rost H, Rosenberger G, Collins BC, Malmström L, et al. Identification of a Set of Conserved Eukaryotic Internal Retention Time Standards for Data-independent Acquisition Mass Spectrometry. Mol Cell Proteomics. 2015; 14(10): 2800-2813.
[8] Li S, Cao Q, Xiao W, Guo Y, Yang Y, Duan X, Shui W. Optimization of Acquisition and Data-Processing Parameters for Improved Proteomic Quantification by Sequential Window Acquisition of All Theoretical Fragment Ion Mass Spectrometry. J Proteome Res. 2017; 16(2):738-747.
[9] Wisniewski, J.R., et al., Universal sample preparation method for proteome analysis. Nat Methods, 2009. 6(5): p.359-62.
声明:本公司出售产品只能用于科研目的,不得用于诊断或者治疗!


